|科技人员揭示组蛋白去甲基化酶参与水稻能量稳态调节表观遗传学机制
科技日报采访人员 刘志伟 通讯员 蒋朝常
【|科技人员揭示组蛋白去甲基化酶参与水稻能量稳态调节表观遗传学机制】9月10日采访人员从华中农业大学获悉 , 该校作物遗传改良国家重点实验室周道绣教授课题组揭示了组蛋白去甲基化酶JMJ705参与水稻能量稳态调节的相关表观遗传学机制 。 相关研究论文近期在《植物细胞》(The Plant Cell )杂志在线发表 。
能量稳态对于所有生命都至关重要 。 与动物相比 , 植物需要更为复杂的适应机制以应对不断变化的环境 , 而能量稳态调节作为环境适应最为重要的一环 , 涉及非常复杂的调控通路 。 真核生物含有一类在进化上极其保守的激酶复合体SNF1 , 其在能量稳态的调节中扮演着核心调控因子的角色 , 这类激酶在植物中被称之为SnRK1 。 SnRK1一般通过对其他蛋白进行磷酸化修饰来进行能量相关信号的传导与调控 。 当能量供应不足时 , SnRK1可以调节相关基因的转录来促进植物能量生成 , 降低能量消耗以维持能量供需平衡 。 早期研究发现SnRK1可以直接结合染色质 , 然而SnRK1如何作用于染色质 , 进而参与基因表达的调控机制仍不清楚 。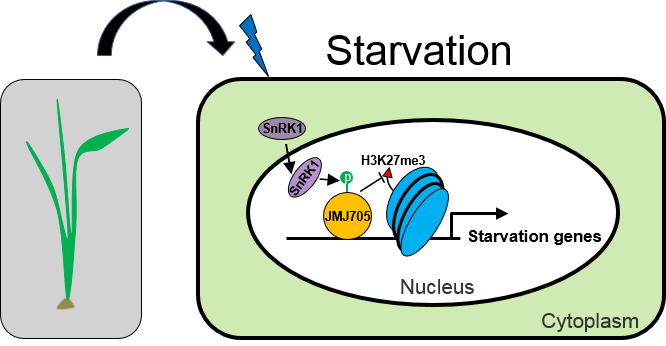
文章插图
该研究发现 , 当水稻处于饥饿状态时 , 饥饿信号促使SnRK1在细胞核中富集 , 与组蛋白H3K27me3去甲基化酶JMJ705发生相互作用 , 并对JMJ705进行磷酸化修饰 , 提高其去除H3K27me3修饰的能力 , 降低饥饿应答基因座位上的H3K27me3修饰水平 , 从而促进这些基因的表达 , 维持细胞中足够的能量供应;在能量充足的情况下 , SnRK1与JMJ705协同抑制饥饿诱导基因的表达 , 减少植物对能量的消耗以保证正常生长发育 。 该研究鉴定的SnRK1-JMJ705通路在植物细胞能量调节、染色质修饰以及基因表达调控之间建立起了直接的联系 , 加深了人们对作物生长发育与逆境应答之间平衡关系的认识 , 对于提高作物产量与环境适应性研究有重要的启发意义 。
华中农业大学生命科学技术学院、作物遗传改良国家重点实验室和湖北洪山实验室周道绣教授课题组博士生王文韬作为该文第一作者 , 周道绣教授为文章通讯作者 , 赵毓教授也参与了该项研究 。
- 俄罗斯联邦|黑客组织REvil成员全部被逮捕
- meta|运用好Facebook组群可以带来哪些好处呢?
- 俄罗斯突袭加密货币勒索组织 REvil,缴获超 600 万美元现金
- 杨植麟:选择创业,是为了从「组织架构」上解决AI落地难题
- revil|俄罗斯突袭加密货币勒索组织 REvil,缴获超 600 万美元现金
- 黑客|闻名世界的黑客组织REvil被抓获:现场缴获大量现金
- 明明是百兆、千兆的宽带|全省首个“全光Wi-Fi”组网服务在株发布
- IT之家1月14日消息|netusbusb共享组件中存在严重的远程代码执行漏洞
- MySQL|mysql 分组查询和聚焦函数,教你更高级—DBA技能包04
- 红米K50细节曝光:侧面指纹+三角形模组设计